AlertsNet2.0
Inzidenz von Blutstrominfektionen, Antibiotikaresistenzen, Blutkulturanforderungen und die Praxis der Blutkulturdiagnostik in Thüringen – ein prospektives populationsbasiertes Register
Das bisherige Scheitern neuer Ansätze zur Behandlung von Blutstrominfektionen (BSI) ist eng mit den Defiziten einer differenzierten Diagnosemöglichkeit verbunden. Der Zeitpunkt der Diagnose und damit die frühzeitige Initiierung therapeutischer Maßnahmen ist jedoch die entscheidende Determinante der hohen Letalität. Dabei ist der Nachweis von Mikroorganismen durch die Blutkultur (BK) für eine adäquate antimikrobielle Therapie der Sepsis unabdingbare Voraussetzung. Erst die Kenntnis des Erregers und seiner Antibiotika-Empfindlichkeit erlaubt nach Einleitung einer empirischen Initialtherapie gezielte antiinfektive Maßnahmen und stellt die Weichen für das weitere diagnostische Vorgehen. Dies verbessert die Prognose, verkürzt die Liegedauer und hilft, eine antiinfektive Übertherapie zu vermeiden.
Die Umsetzung der Blutkulturdiagnostik nach aktuellen Leitlinien ist daher eine wichtige Voraussetzung für eine erfolgreiche Therapie septischer Patienten. Bei lediglich 10 % der Patienten mit schwerer Sepsis gelingt ein Erregernachweis. Lange Transportzeiten der Blutkulturflaschen von der Intensivstation zum mikrobiologischen Labor tragen dazu entscheidend bei. Weiterhin ist aber die Zahl der entnommenen Blutkulturen zu gering (16,3 BK-Sets auf 1.000 Patiententage in Deutschland1). Nach einer Auswertung von Daten des Krankenhaus-Infektions-Surveillance-Systems (KISS) von 223 deutschen Intensivstationen, sollten auf Intensivstationen zumindest 90 BK-Sets/1.000 Patientenliegetage entnommen werden.2 Für Deutschland liegen zudem kaum populationsbezogene Daten zur Antibiotikaresistenz vor und klinische patientenbezogene Daten zu demographischen Faktoren, Morbidität und Letalität sind nicht verfügbar. Dagegen liegen populationsbasierte Daten zu BSI-Raten mit Patientenbezug aus Erhebungen der International Bacteremia Surveillance Collaborative (IBSC)3 u. a. aus Kanada, Skandinavien und Australien vor.
Das thüringenweite prospektive populationsbasierte Register AlertsNet dient dem Aufbau eines Datenerfassungs- und Warnsystems für die Diagnose von BSI, der Identifizierung von Risikofaktoren und der Verbesserung der Behandlung und des Outcomes stationärer Patienten mit BSI. Das Projekt nutzt eine landesweit etablierte elektronische Datenbank für Blutkulturbefunde. Die Datenbank beinhaltet neben diesen Informationen klinische und demographische Daten von Patienten mit nachgewiesener BSI. Von ~316,000 anfallenden BK-Sets/Jahr werden ~25,300 positive BK-Set/Jahr erwartet, von denen wiederum ca. ~20,200 klinisch relevant-positiv sein werden, das entspricht ca. 5.000 Patienten. Primäre Endpunkte sind die Inzidenzrate positiver Blutkulturen (Zahl/1.000 BK-Sets) und die Gesamtzahl entnommener BK-Sets (Zahl/1.000 Patiententage und einsendender Institution).
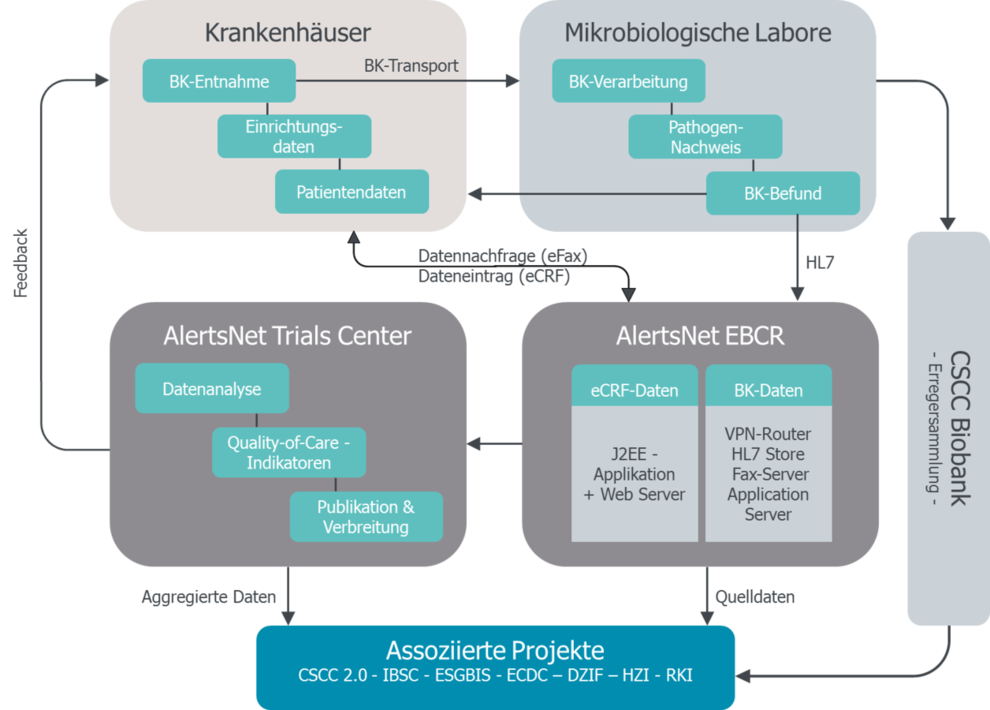
Die erhobenen Daten ermöglichen die Entwicklung von Qualitätsindikatoren und die Bestimmung von Referenzwerten für diese Indikatoren nach Adjustierung der Krankenhauscharakteristika sowie eine Analyse der Beziehung zwischen der Inzidenzrate der Blutkulturentnahme, des Case-Mix, der Fallschwere und Größe der klinischen Einrichtung und der Letalität.
Das Netzwerk wird bis 2018 bis zu 38 Krankenhäuser und 18 mikrobiologische Labore vereinen und somit populationsbezogene Daten für den Freistaat liefern. Eine Übermittlung der Daten an EARS und ein Austausch mit der IBSC ist vorgesehen.
- EARS-Report 2013/2014, European Antimicrobial Resistance Surveillance System, www.ecdc.europa.eu
- Karch A, Castell S, Schwab F, Geffers C, Bongartz H, Brunkhorst FM, Gastmeier P, Mikolajczyk RT: Proposing an empirically justified reference threshold for blood culture sampling rates in intensive care units. J Clin Microbiol 2015; 53(2): 648–52
- Laupland KB, Schønheyder HC, Kennedy KJ, Lyytikäinen O, Valiquette L, Galbraith J, Collignon P, Church DL, Gregson DB, Kibsey P: Rationale for and protocol of a multi-national population-based bacteremia surveillance collaborative. BMC research notes 2009; 2: 146