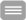Multiplex Ligation-dependent Probe Amplification (MLPA)
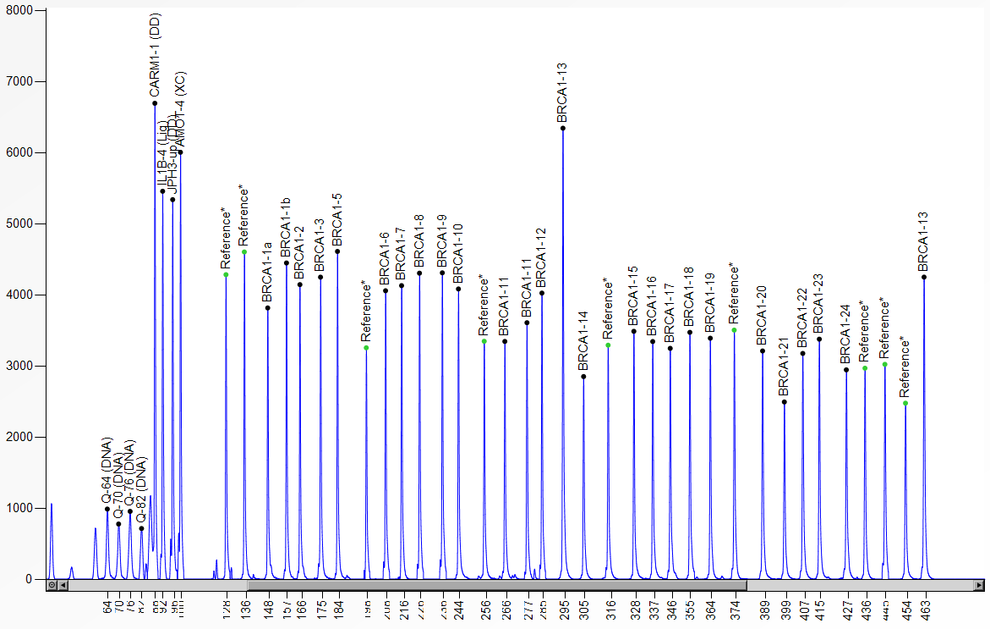
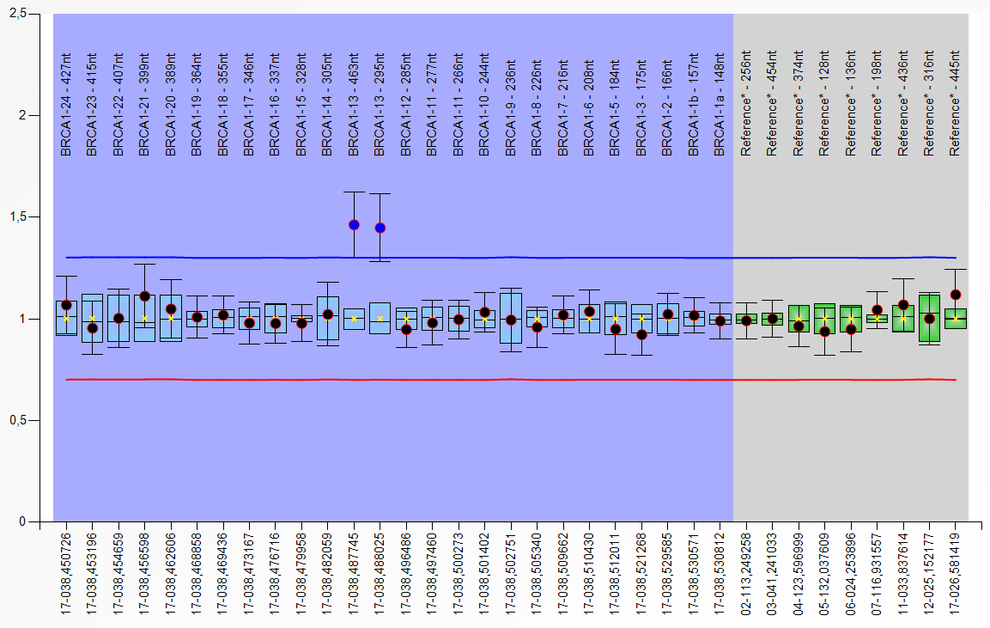
Diese Methode zur Detektion von Kopienzahlveränderungen (Deletionen/ Duplikationen) in nur einem Ansatz mit bis zu 50 verschiedenen DNA-Fragmenten wurde erstmals 2002 beschrieben (Schouten et al., Nucl Acids Res 30:12e57). Die MLPA-Reaktion erfolgt im Wesentlichen in vier Schritten: (1) DNA-Denaturierung und Hybridisierung der MLPA-Proben, (2) Ligationsreaktion, (3) PCR und (4) Trennung der Amplifikationsprodukte durch Kapillarelektrophorese. Kopienzahlveränderungen sind durch Vergrößerung bzw. Reduktion der Peakhöhen/-flächen im Vergleich zu den Kontrollen erkennbar. Jede der bis zu 50 MLPA-Proben in einem Ansatz besteht aus einer “Target”-spezifischen Sequenz, einer “Stuffer”-Sequenz definierter Länge und einer M13-Universal-Primersequenz. Die Multiplex-Amplifikation mit einem Fluoreszenz-markierten M13-Primerpaar kann nur nach Ligation der hybridisierten Proben erfolgen. Durch das Design der unterschiedlich langen “Stuffer”-Fragmente ist eine Trennung von Fragmenten einer Länge zwischen 130 und 480 bp möglich.
Coffalyzer-Auswerteprogramm, MRC Holland
Kommerziell verfügbar sind zahlreiche Kits zur Detektion von Kopienzahlveränderungen ganzer Gene, spezifischer Exons oder Chromosomenbereiche. Auch methylierungsspezifische MLPAs (MS-MLPA) zur gleichzeitigen Detektion von Kopienzahl- und Methylierungsveränderungen sind erhältlich. In unserem Haus kommen zahlreiche Kit’s für die unterschiedlichsten Fragestellungen zum Einsatz (s. Begleitschein mit *), wodurch vor allem auch in Kombination mit der NGS eine weitestgehend vollständige Genanalyse erfolgt.
- Limitierungen:
- Es kann nur eine Aussage über den Primer-abdeckenden bzw. –umspannenden Bereich in Abhängigkeit des verwendeten Kits getroffen werden.
- Das Analyseergebnis kann durch SNPs (Single Nukleotide Polymorphism) in der Primerbindestelle beeinflusst werden.
Literatur: https://www.mlpa.com