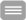Jena (vdG/UKJ). Als rote Punkte im Virusstammbaum sind die 40 aus Thüringen stammenden Proben bezeichnet, die die Arbeitsgruppe von Dr. Christian Brandt mit Bezug zu knapp 10.000 vollständig sequenzierten Genomen des SARS-CoV-2 untersuchten. Das Bioinformatikteam im Institut für Infektionsmedizin und Krankenhaushygiene am Universitätsklinikum Jena nutzte dafür eine repräsentative Auswahl deutscher, europäischer und nichteuropäischer Genomsequenzen, die aus den etwa 300.000 weltweit bekannten und von Wissenschaftler online zur Verfügung gestellten Daten entstammen.
Das Genom von SARS-CoV-2 umfasst etwa 30.000 Basen und ist damit das größte bekannte Genom aller RNA-Viren. Seine molekulare Uhr tickt mit einer Geschwindigkeit von etwa 23 Mutationen pro Jahr - das sind die zufälligen Fehler beim ständigen Kopieren der Virus-Erbinformation, die nicht von vornherein aussortiert werden, weil sie die Zelle absterben lassen. „Das Virus verändert sich, wie es statistisch zu erwarten ist: Es mutiert fröhlich vor sich hin“, fasst Christian Brandt das Bild zusammen, dass sich anhand der untersuchten Stichproben ergibt. Die Proben aus Deutschland lassen sich acht Hauptlinien zuordnen, von denen vier auch in Thüringen nachgewiesen wurden. Die in Großbritannien, Brasilien oder Südafrika verbreiteten Mutationslinien, für die ein höheres Ansteckungspotential bzw. schwächere Immunantworten vermutet werden, sind bislang nicht darunter – noch.