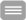humane Nierenorganoide
Insbesondere in der Medikamentenentwicklung zeigen Substanzen häufig unerwartete nephrotoxische Wirkungen. Folglich besteht eine ständige Nachfrage nach genaueren Modellsystemen. Die Verwendung von Nierenorganoiden zur Simulation von Reaktionen auf nephrotoxische Intervention hat das Potenzial, die Lücke zwischen präklinischen Studien zur Wirksamkeit von Medikamenten in Zellkulturen und Tiermodellen und den Stadien klinischer Studien am Menschen zu schließen.
Bild: Jasmin Dilz
Genom Editierung
In der Experimentellen Nephrologie sind verschiedene Systeme etabliert, die es ermöglichen kontrolliert Zielgene in humane Zellenzu integrieren oder auszuschalten oder um Reportergene zu integrieren. So verwenden wir verschiedene Varianten des CRISPR-Cas Systems (Cas9, Cas12, SAM). Auch Lentiviren kommen als genomeditierendes Werkzeug zum Einsatz.
Mit Hilfe der Genom Editierung designen wir unter anderem projektspezifische zellbasierte Assays. Grundlage der Assays sind humane Luciferase-Reporterzellen, welche im Genom ein Lucifrasegen tragen. Das Enzym Luciferase wird ins Cytoplasma freigesetzt und katalysiert in Anwesenheit des Substrates Luciferin eine chemische Reaktion, bei der Licht der Wellenlänge 562 nm (gelb/grün) entsteht. Dieses Lichtsignal kann im lebenden Zustand der Zellen in einem Luminometer detektiert werden. Das gemessene Biolumineszenzsignal korreliert mit der Aktivität der Luciferase und gibt Aufschluss über die Aktivität des untersuchten Faktors der Immunantwort.
CRISPR-Cas, SAM, Lentiviren, direkte oder indirekte Lumineszenz- Fluoreszenzaktivierung
Renin-Angiotensin-Aldosteron-System (RAAS)

Wir untersuchen mit theoretischen und molekularbiologischen Ansätzen Fragestellungen auf dem Gebiet der Genregulation. Der Fokus liegt dabei auf dem Renin-Angiotensin-Aldosteron System (RAAS), das eine entscheidende Rolle für die Regulation des arteriellen Blutdrucks und den Salz-Wasserhaushalt spielt. Eines unserer Schwerpunktprojekte ist die Regulation von Genen im RAAS. Von Interesse sind dabei Mechanismen der Genregulation auf transkriptioneller Ebene wie auch fein modulierende Einflüsse, die z.B. über microRNA vermittelt werden. Methodisch integrieren wir dabei verschiedene biologische Datenentitäten wie z. B. Gen-Expressionsdaten, DNA-Sequenzinformation, Protein-Protein-Wechselwirkungen und Ontologie-Information. Diese sind geeignet, um mit mathematischen Modellen Voraussagen zum Beispiel über die Wirkung von Transkriptionsfaktoren und deren Interaktion zu treffen, die wir dann mit zellbiologischen Experimenten überprüfen. Die enge Kombination von mathematischer Modellierung und experimentellen Ansätzen unterstützt damit die Erforschung von Herz-Kreislauf-Erkrankungen, die die Haupttodesursache in Europa darstellen.
Bild: Kirchner S, Reuter S, Westphal A, Mrowka R. Decipher the complexity of cis-regulatory regions by a modified Cas9. PLoS One. 2020 Jul 2;15(7):e0235530. doi: 10.1371/journal.pone.0235530. PMID: 32614871; PMCID: PMC7332081.
Posttranslationale Genregulation
Mechanismen der Genregulation auf transkriptioneller Ebene wie auch fein modulierende Einflüsse, die z.B. über microRNA vermittelt werden. Methodisch integrieren wir dabei verschiedene biologische Datenentitäten wie z. B. Gen-Expressionsdaten, DNA-Sequenzinformation, Protein-Protein-Wechselwirkungen und Ontologie-Information. Diese sind geeignet, um mit mathematischen Modellen Voraussagen zum Beispiel über die Wirkung von Transkriptionsfaktoren und deren Interaktion zu treffen, die wir dann mit zellbiologischen Experimenten überprüfen. Die enge Kombination von mathematischer Modellierung und experimentellen Ansätzen unterstützt damit die Erforschung von Herz-Kreislauf-Erkrankungen, die die Haupttodesursache in Europa darstellen.
transkriptionelle Isoformen und deren Regulation und Induktion, Deep Sequencing, Cas9-SAM System, CRISPR-Cas Systeme
Regulation der Immunantwort humaner Zellen
Pathogene besitzen auf ihrer Oberfläche Erregergruppen-spezifische molekulare Muster, sogenannte Pathogen-assoziierte molekulare Muster (PAMPs), die von evolutionär konservierten Mustererkennungsrezeptoren (PRRs) der angeborenen Immunität erkannt und gebunden werden. Nach der Bindung einer solchen molekularen Struktur an seinen spezifischen Rezeptor, erfolgt eine Signalweiterleitung bis hin zur Aktivierung verschiedenster Transkriptionsfaktoren, wie nuclear factor 'kappa-light-chain-enhancer' of activated B-cells (NF-κB). Aktiviertes NF-κB transloziert aus dem Cytoplasma in den Zellkern und reguliert dort durch Bindung an spezifische DNA-Motive die Expression einer Vielzahl von bspw. entzündungsfördernden Genen.
Grundlage der zellbasierten Assays sind humane Luciferase-Reporterzellen, welche einen NF-κB sensitiven Promotor im Genom tragen, der ein nachfolgendes Luciferasegen reguliert. Transloziert der aktivierte Transkriptionsfaktor NF-κB aus dem Cytoplasma in den Zellkern, bindet er dort an spezifische Bindemotive in der NF-κB sensitiven Promotorregion. Dadurch wird die Transkription und Translation des folgenden Luciferasegens aktiviert.
TLR Signalwege, Erreger-Host Interaktion,
KI unterstützte Gen-Analysen
Maschinelles Lernen, Deep sequencing analyse
Numerische und experimentelle Analyse
Gen-Regulation, Protein-Protein Interaktionen, Gene-Expressions Daten, Transcriptions Faktoren, miRNA Funktion, substanzvermittelte iPS, TLR Signalweg, Toxizität, Erreger-Host Interaktion
Zeitreihenanalyse
Analyse der Interaktion von Atem- und Herz-Kreislauf-System, EKG-Prädiktoren, T-Wellen Alternans
Modellierung von physiologischen Systemen
mit Schwerpunkt Herz-Kreislauf-System, Renin-Angiotensin-Aldosteron-System