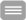AG Optisch-molekulare Diagnostik und Systemtechnologie
>> Forschungsschwerpunkte
- Optische, molekulare und serologische Multiparameteranalysen für die klinisch-mikrobiologische Diagnostik und Epidemiologie
- Schwerpunkt: Detektion und Verständnis von Antibiotikaresistenzen pathogener Bakterien
- Assay-basierte Forschungsaktivitäten mit Hilfe modernster molekularer Technologien, wie digitaler PCR, real-time PCR, Next-Generation-Sequenzierung (NGS), Mikroarray-basierte und isothermale Analyseverfahren
- Mikroarray-basierte Muliparameterserologie mittels Antikörperarrays, Antigenarrays und Peptidarrays
- Bioinformatik für das molekulare Assaydesign und der Auswertung entsprechender Daten (z.B. NGS)
- Translation bestehender und neuer Forschungsergebnisse in real verfügbare Produkte in Kooperation mit entsprechenden Partnern
- Aufbau einer „Brücke“ zwischen akademischer Forschung zur Entwicklung neuer innovativer Assays mittels Systemtechnologie und Produktentwicklung und -realisierung mit kooperierenden Firmen der Diagnostikindustrie
DNS-basierte Multiparameterverfahren
Die Fachabteilung Ehricht hat ihre Arbeit am IPHT im Januar 2019 aufgenommen und vorher, in den vergangenen Jahren, wesentliche Erkenntnisse bei der Entwicklung und Anwendung verschiedenster molekularer Verfahrung für die Detektion, die Epidemiologie und das Verständnis von pathogenen Bakterien und deren Antibiotikaresistenzen (MRSA, ESBL, CRE, VRE u.a.) gewonnen. Dabei kamen vor allem Mikroarray-basierte Verfahren und eigens dafür entwickelte Plattformen zum Einsatz (Abbildung links), die in ihrer gesamten Breite über bioinformatisches Assaydesign, Oberflächenchemie, Fertigung, Testentwicklung, Bild- und Datenauswertung sowie spezifische Anwendungen reichte. Besonderes Augenmerk wurde einerseits auf die Implementierung dieser Forschungsergebnisse in real existierende Produkte und deren Evolution gelegt. Andererseits war ein Hauptfokus, Strategien und Verfahren für die optimale Probenvorbereitung zu erarbeiten und in komplexe Abläufe zu implementieren.
Next-Generation-Sequenzierung – Ein Werkzeug zur Entwicklung von Schnelltests
Die von Oxford Nanopore Technologies (Oxford, UK) entwickelte Plattform MinION wird mit ihrer Porentechnologie als der nächste Schritt der Next-Generation-Sequenzierung betrachtet (Abbíldung rechts). Die MinION Technologie basiert auf membrangebundene Protein-Nanoporen. Liegt eine Spannung an dieser Membran an, wird ein Ionenstrom durch die Poren generiert. Passiert ein DNA-Strang die Pore kommt es zu Spannungsänderungen, welche gemessen und später zu Nukleotiden umgerechnet werden. Je Pore können bis zu 400 Nukleotide pro Sekunde gemessen werden, was pro Genom zu Ausgaben von bis zu zwei Gigabasen führen kann. Einzelne Reads können Längen von 50.000-100.000 Basen erreichen. Daher ist es möglich speziell bakterielle Genome einfach und schnell zu analysieren und zu charakterisieren.
Was die Plattform so attraktiv macht, ist ihr vergleichbar günstiger Preis und die kompakte Größe. Die AG Ehricht will daher diese Technologie nutzen, neue bahnbrechende Assays auf molekularer Basis zu entwickeln. Allerdings werden auch andere Methoden der Next-Generation-Sequenzierung (z.B. MySeq, Illumina) etabliert und angewandt.
Protein- und Peptid-basierte Multiparameterverfahren
Routinemäßig werden Tests für verschiedene Antigene getrennt und nacheinander durchgeführt, was zu hohen Kosten und zeitlich verzögerten Ergebnissen führt. Außerdem wird eine relativ große Menge an Probe benötigt. Die AG Ehricht konnte belegen, dass die Multiparameter-Serologie mit Protein- und Peptidmikroarrays realisierbar ist. So konnte der IgG-Status und Impfstatus für 30 Erreger parallel und aus 1 µl und einer Gesamtzeit von 2h bestimmt werden (Abbildung unten). Das gleiche Verfahren könnte in Zukunft nicht nur für diagnostische Zwecke, sondern auch für die Testentwicklung (Antigen- und Antikörperscreening) eingesetzt werden. Das Panel der Antigene kann schnell an andere Anwendungen angepasst werden und auch auf Point-of-Care Plattformen wie Lateral Flow übertragen werden.
>> Projekte
Aktuelle Projekte
RESISTOVAC Rapid and economical POC tests for determination of immune status and bacterial resistance factors
ADA Adaptierbare dezentrale Diagnostik für die Tier- und Humanmedizin
AVATAR Anonymisierung persönlicher Gesundheitsdaten durch Erzeugung digitaler Avatare in Medizin und Pflege
VRE-DETEKT Molekulare Mikroarrayplattform zur raschen Erkennung und Charakterisierung vancomycin-resistenter Enterokokken
RESISTOVAC Testformate zur schnellen Erfassung des Impfstatus
SANGUINE Diagnostik von Blut- und Knochenmarkkrebs
AMPEL „Untersuchung synergistischer Effekte von membran-aktiven Peptiden und klassischen Antibiotika
frühere Projekte:
ResiCheck Test-Entwicklung zum Nachweis modifizierter Penizillin Bindeproteine in klinischen MRSA-Isolaten
DRESI Schnelle und präzise Diagnostik und Resistenzprüfung von Erregern der Sepsis auf der Intensivstation
DIGLEBEN Digitalisierung der Lebenswissenschaften
MULTIHOLODIAG Multiparameter-Analysen mit optisch-holografischen Verfahren für die medizinische Diagnostik
Publikationen
2023:
- Szumlanski T, Neumann B, Bertram R, Simbeck A, Ziegler R, Monecke S, Ehricht R, Schneider-Brachert W, Steinmann J. (2023) Characterization of PVL-Positive MRSA Isolates in Northern Bavaria, Germany over an Eight-Year Period. Microorganisms. 2022 Dec 24;11(1):54. doi: 10.3390/microorganisms11010054.
- Monecke S, Bedewy AK, Müller E, Braun SD, Diezel C, Elsheredy A, Kader O, Reinicke M, Ghazal A, Rezk S, Ehricht R. (2023) Characterisation of Methicillin- Resistant Staphylococcus aureus from Alexandria, Egypt. Antibiotics (Basel). 2023 Jan 1;12(1):78. doi: 10.3390/antibiotics12010078.
- Wagner GE, Berner A, Lipp M, Kohler C, Assig K, Lichtenegger S, Saqib M, Müller E, Trinh TT, Gad AM, Söffing HH, Ehricht R, Laroucau K, Steinmetz I. (2023) Protein Microarray-Guided Development of a Highly Sensitive and Specific Dipstick Assay for Glanders Serodiagnostics. J Clin Microbiol. 2023 Jan 26;61(1):e0123422. doi: 10.1128/jcm.01234-22.
- Aloba BK, Kinnevey PM, Monecke S, Brennan GI, O'Connell B, Blomfeldt A, McManus BA, Schneider-Brachert W, Tkadlec J, Ehricht R, Senok A, Bartels MD, Coleman DC. (2023) An emerging Panton-Valentine leukocidin-positive CC5-meticillin- resistant Staphylococcus aureus-IVc clone recovered from hospital and community settings over a 17-year period from 12 countries investigated by whole-genome sequencing. J Hosp Infect. 2023 Feb;132:8-19. doi: 10.1016/j.jhin.2022.11.015
- Wagner GE, Dabernig-Heinz J, Lipp M, Cabal A, Simantzik J, Kohl M, Scheiber M, Lichtenegger S, Ehricht R, Leitner E, Ruppitsch W, Steinmetz I. (2023) Real-Time Nanopore Q20+ Sequencing Enables Extremely Fast and Accurate Core Genome MLST Typing and Democratizes Access to High-Resolution Bacterial Pathogen Surveillance. J Clin Microbiol. 2023 Apr 20;61(4):e0163122. doi: 10.1128/jcm.01631-22.
- Burgold-Voigt S, Monecke S, Busch A, Bocklisch H, Braun SD, Diezel C, Hotzel H, Liebler-Tenorio EM, Müller E, Reinicke M, Reissig A, Ruppelt-Lorz A, Ehricht R. (2023) Characterisation of a <i>Staphylococcus aureus</i> Isolate Carrying Phage-Borne Enterotoxin E from a European Badger (Meles meles). Pathogens. 2023 May 12;12(5):704. doi: 10.3390/pathogens12050704.
- Pahlow S, Richard-Lacroix M, Hornung F, Köse-Vogel N, Mayerhöfer TG, Hniopek J, Ryabchykov O, Bocklitz T, Weber K, Ehricht R, Löffler B, Deinhardt-Emmer S, Popp J. (2023) Simple, Fast and Convenient Magnetic Bead-Based Sample Preparation for Detecting Viruses via Raman-Spectroscopy. Biosensors (Basel). 2023 May 30;13(6):594. doi: 10.3390/bios13060594.
- Monecke S, Akpaka PE, Smith MR, Unakal CG, Thoms Rodriguez CA, Ashraph K, Müller E, Braun SD, Diezel C, Reinicke M, Ehricht R. (2023) Clonal Complexes Distribution of Staphylococcus aureus Isolates from Clinical Samples from the Caribbean Islands. Antibiotics (Basel). 2023 Jun 14;12(6):1050. doi: 10.3390/antibiotics12061050.
- Bhat VJ, Blaschke D, Müller E, Ehricht R, Schmidt H. (2023) A Novel Approach to Monitor the Concentration of Phosphate Buffers in the Range of 1 M to 0.1 M Using a Silicon-Based Impedance Sensor. Biosensors (Basel). 2023 Aug 24;13(9):841. doi: 10.3390/bios13090841.
- Braun SD, Asif M, Hassan L, Ul Haq I, Abbasi SA, Jamil B, Monecke S, Ehricht R, Mueller E, Syed MA.(2023) Analysis of carbapenem-resistant strains isolated in a tertiary care hospital in Rawalpindi, Pakistan, during the years 2016 and 2020. J Hosp Infect. 2023 Nov;141:187-189. doi: 10.1016/j.jhin.2023.06.007.
- Moawad AA, El-Adawy H, Linde J, Jost I, Gärtner T, Hruschka K, Donat K, Neubauer H, Monecke S, Tomaso H (2023) Whole genome sequence-based analysis of Staphylococcus aureus isolated from bovine mastitis in Thuringia, Germany Front. Microbiol., 25 August 2023 Sec. Antimicrobials, Resistance and Chemotherapy, doi: 10.3389/fmicb.2023.1216850
- Osadare IE, Xiong L, Rubio I, Neugebauer U, Press AT, Ramoji A, Popp J. (2023) Raman Spectroscopy Profiling of Splenic T-Cells in Sepsis and Endotoxemia in Mice Int J Mol Sci 2023 Jul 27;24(15):12027. doi: 10.3390/ijms241512027. PMID: 37569403 PMCID: PMC10419286 DOI: 10.3390/ijms241512027
- Triebel S, Sachse K, Weber M, Heller M, Diezel C, Hölzer M, Schnee C, Marz M. (2023) De novo genome assembly resolving repetitive structures enables genomic analysis of 35 European Mycoplasmopsis bovis strains. BMC Genomics 2023 Sep 16;24(1):548. doi: 10.1186/s12864-023-09618-5.
- Loncaric I, Keinprecht H, Irimaso E, Cabal-Rosel A, Stessl B, Ntakirutimana C, Marek L, Fischer O, Szostak M, Oberrauch C, Wittek T, Müller E, Desvars A, Feßler A, Braun S, Schwarz, S, Ehling-Schultz M, Monecke S, Ehricht R, Ruppitsch W, Grunert T, Spergser J. (2023) Diversity of Staphylococcus aureus isolated from nares of ruminants Journal of Applied Microbiology - Decision on Manuscript ID JAMICRO-2023-0994.R1
2022:
- Monecke S, Feßler AT, Burgold-Voigt S, Krüger H, Mühldorfer K, Wibbelt G, Liebler-Tenorio EM, Reinicke M, Braun SD, Hanke D, Diezel C, Müller E, Loncaric I, Schwarz S, Ehricht R. (2021) Staphylococcus aureus isolates from Eurasian Beavers (Castor fiber) carry a novel phage-borne bicomponent leukocidin related to the Panton-Valentine leukocidin. Sci Rep. 2021 Dec 22;11(1):24394. doi: 10.1038/s41598-021-03823-6.
- Tsilipounidaki K, Athanasakopoulou Z, Müller E, Burgold-Voigt S, Florou Z, Braun SD, Monecke S, Gatselis NK, Zachou K, Stefos A, Tsagalas I, Sofia M, Spyrou V, Billinis C, Dalekos GN, Ehricht R, Petinaki E. (2022) Plethora of Resistance Genes in Carbapenem-Resistant Gram-Negative Bacteria in Greece: No End to a Continuous Genetic Evolution. Microorganisms. 2022 Jan 13;10(1):159. doi: 10.3390/microorganisms10010159.
- El-Ashker M, Monecke S, Gwida M, Saad T, El-Gohary A, Mohamed A, Reißig A, Frankenfeld K, Gary D, Müller E, Ehricht R. (2022) Molecular characterisation of methicillin-resistant and methicillin-susceptible Staphylococcus aureus clones isolated from healthy dairy animals and their caretakers in Egypt. Vet Microbiol. 2022 Apr;267:109374. doi: 10.1016/j.vetmic.2022.109374. Epub 2022 Feb
- Luzzago C, Lauzi S, Ehricht R, Monecke S, Corlatti L, Pedrotti L, Piccinini R. Survey of Staphylococcus aureus carriage by free-living red deer (Cervus elaphus): Evidence of human and domestic animal lineages. (2022) Transbound Emerg Dis. 2022 Sep;69(5):e1659-e1669. doi: 10.1111/tbed.14500. Epub 2022 Mar 22.
- Marquet M, Zöllkau J, Pastuschek J, Viehweger A, Schleußner E, Makarewicz O, Pletz MW, Ehricht R, Brandt C. Evaluation of microbiome enrichment and host DNA depletion in human vaginal samples using Oxford Nanopore's adaptive sequencing. (2022) Sci Rep. 2022 Mar 7;12(1):4000. doi: 10.1038/s41598-022-08003-8.
- Finsterwalder SK, Loncaric I, Cabal A, Szostak MP, Barf LM, Marz M, Allerberger F, Burgener IA, Tichy A, Feßler AT, Schwarz S, Monecke S, Ehricht R, Ruppitsch W, Spergser J, Künzel F. (2022) Dogs as carriers of virulent and resistant genotypes of Clostridioides difficile. Zoonoses Public Health. 2022 Sep;69(6):673-681. doi: 10.1111/zph.12956. Epub 2022 May 12. PMID: 35546073;
- Burgold-Voigt S, Müller E, Zopf D, Monecke S, Braun SD, Frankenfeld K, Kiehntopf M, Weis S, Schumacher T, Pletz MW, Ehricht R; CoNAN Study Group. Development of a new antigen-based microarray platform for screening and detection of human IgG antibodies against SARS-CoV-2. (2022) Sci Rep. 2022 May 16;12(1):8067. doi: 10.1038/s41598-022-10823-7.
- Monecke S, Schaumburg F, Shittu AO, Schwarz S, Mühldorfer K, Brandt C, Braun SD, Collatz M, Diezel C, Gawlik D, Hanke D, Hotzel H, Müller E, Reinicke M, Feßler AT, Ehricht R. (2022) Description of Staphylococcal Strains from Straw-Coloured Fruit Bat (Eidolon helvum) and Diamond Firetail (Stagonopleura guttata) and a Review of their Phylogenetic Relationships to Other Staphylococci. Front Cell Infect Microbiol. 2022 May 11;12:878137. doi: 10.3389/fcimb.2022.878137.
- Tsekouras N, Athanasakopoulou Z, Diezel C, Kostoulas P, Braun SD, Sofia M, Monecke S, Ehricht R, Chatzopoulos DC, Gary D, Krähmer D, Spyrou V, Christodoulopoulos G, Billinis C, Papatsiros VG. (2022) Cross-Sectional Survey of Antibiotic Resistance in Extended Spectrum β-Lactamase-Producing Enterobacteriaceae Isolated from Pigs in Greece. (2022) Animals (Basel). 2022 Jun 16;12(12):1560. doi: 10.3390/ani12121560.
- Athanasakopoulou Z, Diezel C, Braun SD, Sofia M, Giannakopoulos A, Monecke S, Gary D, Krähmer D, Chatzopoulos DC, Touloudi A, Birtsas P, Palli M, Georgakopoulos G, Spyrou V, Petinaki E, Ehricht R, Billinis C. (2022) Occurrence and Characteristics of ESBL- and Carbapenemase- Producing Escherichia coli from Wild and Feral Birds in Greece. Microorganisms. 2022 Jun 14;10(6):1217. doi: 10.3390/microorganisms10061217.
- Pistiki A, Monecke S, Shen H, Ryabchykov O, Bocklitz TW, Rösch P, Ehricht R, Popp J. (2022) Comparison of Different Label-Free Raman Spectroscopy Approaches for the Discrimination of Clinical MRSA and MSSA Isolates. (2022) Microbiol Spectr. 2022 Oct 26;10(5):e0076322. doi: 10.1128/spectrum.00763-22. Epub 2022 Aug 25.
- Moawad AA, Hotzel H, Hafez HM, Ramadan H, Tomaso H, Braun SD, Ehricht R, Diezel C, Gary D, Engelmann I, Zakaria IM, Reda RM, Eid S, Shahien MA, Neubauer H, Monecke S. (2022) Occurrence, Phenotypic and Molecular Characteristics of Extended- Spectrum Beta-Lactamase-Producing Escherichia coli in Healthy Turkeys in Northern Egypt. Antibiotics (Basel). 2022 Aug 8;11(8):1075. doi: 10.3390/antibiotics11081075.
- Monecke S, Roberts MC, Braun SD, Diezel C, Müller E, Reinicke M, Linde J, Joshi PR, Paudel S, Acharya M, Chalise MK, Feßler AT, Hotzel H, Khanal L, Koju NP, Schwarz S, Kyes RC, Ehricht R. (2022) Sequence Analysis of Novel Staphylococcus aureus Lineages from Wild and Captive Macaques. (2022) Int J Mol Sci. 2022 Sep 23;23(19):11225. doi: 10.3390/ijms231911225.
- Steinig E, Aglua I, Duchene S, Meehan MT, Yoannes M, Firth C, Jaworski J, Drekore J, Urakoko B, Poka H, Wurr C, Ebos E, Nangen D, Müller E, Mulvey P, Jackson C, Blomfeldt A, Aamot HV, Laman M, Manning L, Earls M, Coleman DC, Greenhill A, Ford R, Stegger M, Syed MA, Jamil B, Monecke S, Ehricht R, Smith S, Pomat W, Horwood P, Tong SYC, McBryde E. (2022) Phylodynamic signatures in the emergence of community-associated MRSA. Proc Natl Acad Sci U S A. 2022 Nov 8;119(45):e2204993119. doi: 10.1073/pnas.2204993119. Epub 2022 Nov 2.
- Marquet M, Hölzer M, Pletz MW, Viehweger A, Makarewicz O, Ehricht R, Brandt C. (2022) What the Phage: a scalable workflow for the identification and analysis of phage sequences. Gigascience. 2022 Nov 18;11:giac110. doi: 10.1093/gigascience/giac110.
- Aloba BK, Kinnevey PM, Monecke S, Brennan GI, O'Connell B, Blomfeldt A, McManus BA, Schneider-Brachert W, Tkadlec J, Ehricht R, Senok A, Bartels MD, Coleman DC. (2022) An emerging Panton-Valentine leukocidin (PVL)-positive CC5-meticillin-resistant Staphylococcus aureus-IVc clone recovered from hospital and community settings over a 17-year period from 12 countries investigated by whole-genome sequencing. J Hosp Infect. 2022 Dec 5:S0195-6701(22)00369-3. doi: 10.1016/j.jhin.2022.11.015. Epub ahead of print.
- Collatz M, Braun SD, Monecke S and Ehricht R (2022); ConsensusPrime—A Bioinformatic Pipeline for Ideal Consensus Primer Design.; BioMedInformatics 2022, 2(4), 637-642; doi: 10.3390/biomedinformatics2040041
- Akarsu H, Liljander A, Younan M, Brodard I, Overesch G, Glücks I, Labroussaa F, Kuhnert P, Perreten V, Monecke S, Drexler JF, Corman VM, Falquet L, Jores J. (2022) Genomic Characterization and Antimicrobial Susceptibility of Dromedary- Associated Staphylococcaceae from the Horn of Africa. Appl Environ Microbiol. 2022 Nov 8;88(21):e0114622. doi: 10.1128/aem.01146-22. Epub 2022 Oct 13.
- Engler C, Renna MS, Beccaria C, Silvestrini P, Pirola SI, Pereyra EAL, Baravalle C, Camussone CM, Monecke S, Calvinho LF, Dallard BE. Differential immune response to two Staphylococcus aureus strains with distinct adaptation genotypes after experimental intramammary infection of dairy cows. Microb Pathog. 2022 Nov;172:105789. doi: 10.1016/j.micpath.2022.105789. Epub 2022 Sep 20.
2021:
- Ibadan, Nigeria. Fashae K, Engelmann I, Monecke S, Braun SD, Ehricht R. Molecular characterisation of extended-spectrum ß-lactamase producing Escherichia coli in wild birds and cattle, BMC Vet Res. 2021 Jan 18;17(1):33. doi: 10.1186/s12917-020-02734-4. PMID: 33461554
- McManus BA, Aloba BK, Earls MR, Brennan GI, O'Connell B, Monecke S, Ehricht R, Shore AC, Coleman DC. Multiple distinct outbreaks of Panton-Valentine leucocidin-positive community-associated meticillin-resistant Staphylococcus aureus in Ireland investigated by whole-genome sequencing. J Hosp Infect. 2021 Feb;108:72-80. doi: 10.1016/j.jhin.2020.11.021. Epub 2020 Nov 28. PMID: 33259881
- Bernreiter-Hofer T, Schwarz L, Müller E, Cabal-Rosel A, Korus M, Misic D, Frankenfeld K, Abraham K, Grünzweil O, Weiss A, Feßler AT, Allerberger F, Schwarz S, Szostak MP, Ruppitsch W, Ladinig A, Spergser J, Braun SD, Monecke S, Ehricht R, Loncaric I. The Pheno- and Genotypic Characterization of Porcine Escherichia coli Isolates. 2021 Aug 6;9(8):1676. doi: 10.3390/microorganisms9081676. PMID: 34442755
- Grünzweil OM, Palmer L, Cabal A, Szostak MP, Ruppitsch W, Kornschober C, Korus M, Misic D, Bernreiter-Hofer T, Korath ADJ, Feßler AT, Allerberger F, Schwarz S, Spergser J, Müller E, Braun SD, Monecke S, Ehricht R, Walzer C, Smodlaka H, Loncaric I. Presence of β-Lactamase-producing Enterobacterales and Salmonella Isolates in Marine Mammals. Int J Mol Sci. 2021 May 31;22(11):5905. doi: 10.3390/ijms22115905. PMID: 34072783
- Borg MA, Monecke S, Haider J, Müller E, Reissig A, Ehricht R. MRSA improvement within a highly endemic hospital in Malta: infection control measures or clonal change? J Hosp Infect. 2021 Apr; 110:201-202. doi: 10.1016/j.jhin.2021.02.003. Epub 2021 Feb 9. PMID: 33577835 No abstract available.
- Mišić D, Kiskaroly F, Szostak MP, Cabal A, Ruppitsch W, Bernreiter-Hofer T, Milovanovic V, Feßler AT, Allerberger F, Spergser J, Müller E, Schwarz S, Braun SD, Monecke S, Ehricht R, Korus M, Benković D, Korzeniowska M, Loncaric I. The First Report of mcr-1-Carrying Escherichia coli Originating from Animals in Serbi Antibiotics (Basel). 2021 Sep 3;10(9):1063. doi: 10.3390/antibiotics10091063. PMID: 34572647
- Athanasakopoulou Z, Reinicke M, Diezel C, Sofia M, Chatzopoulos DC, Braun SD, Reissig A, Spyrou V, Monecke S, Ehricht R, Tsilipounidaki K, Giannakopoulos A, Petinaki E, Billinis C. Antimicrobial Resistance Genes in ESBL-Producing Escherichia coli Isolates from Animals in Greece. Antibiotics (Basel). 2021 Apr 4;10(4):389. doi: 10.3390/antibiotics10040389. PMID: 33916633 Free PMC article.
- Achek R, El-Adawy H, Hotzel H, Hendam A, Tomaso H, Ehricht R, Neubauer H, Nabi I, Hamdi TM, Monecke S. Molecular Characterization of Staphylococcus aureus Isolated from Human and Food Samples in Northern Algeria. 2021 Oct 3;10(10):1276. doi: 10.3390/pathogens10101276. PMID: 34684225
- Senok A, Monecke S, Nassar R, Celiloglu H, Thyagarajan S, Müller E, Ehricht R. Lateral Flow Immunoassay for the Detection of Panton-Valentine Leukocidin in Staphylococcus aureus From Skin and Soft Tissue Infections in the United Arab Emirates. Front Cell Infect Microbiol. 2021 Oct 18;11:754523. doi: 10.3389/fcimb.2021.754523. eCollection 2021. PMID: 34733796
- Madzgalla S, Duering H, Hey JC, Neubauer S, Feller KH, Ehricht R, Pletz MW, Makarewicz O. Assessment of Phenotype Relevant Amino Acid Residues in TEM-β-Lactamases by Mathematical Modelling and Experimental Approval. 2021 Aug 13;9(8):1726. doi: 10.3390/microorganisms9081726. PMID: 34442804
- Monecke S, Müller E, Braun SD, Armengol-Porta M, Bes M, Boswihi S, El-Ashker M, Engelmann I, Gawlik D, Gwida M, Hotzel H, Nassar R, Reissig A, Ruppelt-Lorz A, Senok A, Somily AM, Udo EE, Ehricht R. Characterisation of S. aureus/MRSA CC1153 and review of mobile genetic elements carrying the fusidic acid resistance gene fusC. Sci Rep. 2021 Apr 14;11(1):8128. doi: 10.1038/s41598-021-86273-4. PMID: 33854075
- Sarkhoo E, Udo EE, Boswihi SS, Monecke S, Mueller E, Ehricht R. The Dissemination and Molecular Characterization of Clonal Complex 361 (CC361) Methicillin-Resistant Staphylococcus aureus (MRSA) in Kuwait Hospitals. Front Microbiol. 2021 May 6;12:658772. doi: 10.3389/fmicb.2021.658772. eCollection 2021. PMID: 34025612
- Earls MR, Steinig EJ, Monecke S, Samaniego Castruita JA, Simbeck A, Schneider-Brachert W, Vremerǎ T, Dorneanu OS, Loncaric I, Bes M, Lacoma A, Prat Aymerich C, Wernery U, Armengol-Porta M, Blomfeldt A, Duchene S, Bartels MD, Ehricht R, Coleman DC. Exploring the evolution and epidemiology of European CC1-MRSA-IV: tracking a multidrug-resistant community-associated meticillin-resistant Staphylococcus aureus clone. Microb Genom. 2021 Jul;7(7):000601. doi: 10.1099/mgen.0.000601. PMID: 34223815
- . Sáez Moreno D, Visram Z, Mutti M, Restrepo-Córdoba M, Hartmann S, Kremers AI, Tišáková L, Schertler S, Wittmann J, Kalali B, Monecke S, Ehricht R, Resch G, Corsini L. ε2-Phages Are Naturally Bred and Have a Vastly Improved Host Range in Staphylococcus aureus over Wild Type Phages Pharmaceuticals (Basel). 2021 Apr 2;14(4):325. doi: 10.3390/ph14040325. PMID: 33918287
- Schauer B, Szostak MP, Ehricht R, Monecke S, Feßler AT, Schwarz S, Spergser J, Krametter-Frötscher R, Loncaric I. Diversity of methicillin-resistant coagulase-negative Staphylococcus spp. and methicillin-resistant Mammaliicoccus spp. isolated from ruminants and New World camelids. Vet Microbiol. 2021 Mar;254:109005. doi: 10.1016/j.vetmic.2021.109005. Epub 2021 Feb 4. PMID: 33582485
- Thunberg U, Hugosson S, Ehricht R, Monecke S, Müller E, Cao Y, Stegger M, Söderquist B. Long-Term Sinonasal Carriage of Staphylococcus aureus and Anti-Staphylococcal Humoral Immune Response in Patients with Chronic Rhinosinusitis. 2021 Jan 27;9(2):256. doi: 10.3390/microorganisms9020256. PMID: 33513900
- Christ B, Collatz M, Dahmen U, Herrmann K-H, Höpfl S, König M, Lambers L, Marz M, Meyer D, Radde N, Reichenbach JR, Ricken T and Tautenhahn H-M Hepatectomy-Induced Alterations in Hepatic Perfusion and Function - Toward Multi-Scale Computational Modeling for a Better Prediction of Post-hepatectomy Liver Function. (2021) Physiol. 12:733868. doi: 10.3389/fphys.2021.733868
- Burgold-Voigt S, Monecke S, Simbeck A, Holzmann T, Kieninger B, Liebler-Tenorio EM, Braun SD, Collatz M, Diezel C, Müller E, Schneider-Brachert W and Ehricht R Characterisation and Molecular Analysis of an Unusual Chimeric Methicillin Resistant Staphylococcus Aureus Strain and its Bacteriophages. (2021) Genet. 12:723958. doi: 10.3389/fgene.2021.723958
- Ahmed W, Neubauer H, Tomaso H, El Hofy FI, Monecke S, Abd El-Tawab AA, Hotzel H. Characterization of Enterococci- and ESBL-Producing Escherichia coli Isolated from Milk of Bovides with Mastitis in Egypt. 2021 Jan 21;10(2):97. doi: 10.3390/pathogens10020097. PMID: 33494211
2020:
- Antók FI, Mayrhofer R, Marbach H, Masengesho JC, Keinprecht H, Nyirimbuga V, Fischer O, Lepuschitz S, Ruppitsch W, Ehling-Schulz M, Feßler AT, Schwarz S, Monecke S, Ehricht R, Grunert T, Spergser J, Loncaric I. (2020) Characterization of Antibiotic and Biocide Resistance Genes and Virulence Factors of Staphylococcus Species Associated with Bovine Mastitis in Rwanda. Antibiotics (Basel). 2019 Dec 18;9(1):1. doi: 10.3390/antibiotics9010001
- Gwida M, Awad A, El-Ashker M, Hotzel H, Monecke S, Ehricht R, Müller E, Reißig A, Barth SA, Berens C, Braun SD. (2020) Microarray-based detection of resistance and virulence factors in commensal Escherichia coli from livestock and farmers in Egypt. Vet Microbiol. 2020 Jan;240:108539. doi: 10.1016/j.vetmic.2019.108539
- El-Ashker M, Gwida M, Monecke S, El-Gohary F, Ehricht R, Elsayed M, Akinduti P, El-Fateh M, Maurischat S. (2020) Antimicrobial resistance pattern and virulence profile of S. aureus isolated from household cattle and buffalo with mastitis in Egypt. Vet Microbiol. 2020 Jan;240:108535. doi: 10.1016/j.vetmic.2019.108535
- Senok A, Nassar R, Kaklamanos EG, Belhoul K, Abu Fanas S, Nassar M, Azar AJ, Müller E, Reissig A, Gawlik D, Monecke S, Ehricht R. (2020) Molecular Characterization of Staphylococcus aureus Isolates Associated with Nasal Colonization and Environmental Contamination in Academic Dental Clinics. Microb Drug Resist. 2020 Jun;26(6):661-669. doi: 10.1089/mdr.2019.0318
- Achek R, Hotzel H, Nabi I, Kechida S, Mami D, Didouh N, Tomaso H, Neubauer H, Ehricht R, Monecke S, El-Adawy H. (2020)Phenotypic and Molecular Detection of Biofilm Formation in Staphylococcus aureus Isolated from Different Sources in Algeria. Pathogens. 2020 Feb 24;9(2):153. doi: 10.3390/pathogens9020153
- Loreck K, Mitrenga S, Heinze R, Ehricht R, Engemann C, Lueken C, Ploetz M, Greiner M, Meemken D. (2020) Use of meat juice and blood serum with a miniaturised protein microarray assay to develop a multi-parameter IgG screening test with high sample throughput potential for slaughtering pigs. BMC Vet Res. 2020 Apr 6;16(1):106. doi: 10.1186/s12917-020-02308-4
- Rödel J, Edel B, Braun SD, Ehricht R, Simon S, Fruth A, Löffler B. (2020) Simple differentiation of Salmonella Typhi, Paratyphi and Choleraesuis from Salmonella species using the eazyplex TyphiTyper LAMP assay. J Med Microbiol. 2020 Jun;69(6):817-823. doi: 10.1099/jmm.0.001201
- Monecke S, König E, Earls MR, Leitner E, Müller E, Wagner GE, Poitz DM, Jatzwauk L, Vremerǎ T, Dorneanu OS, Simbeck A, Ambrosch A, Zollner-Schwetz I, Krause R, Ruppitsch W, Schneider-Brachert W, Coleman DC, Steinmetz I, Ehricht R. (2020) An epidemic CC1-MRSA-IV clone yields false-negative test results in molecular MRSA identification assays: a note of caution, Austria, Germany, Ireland, 2020. Euro Surveill. 2020;25(25):2000929. doi: 10.2807/1560-7917.ES.2020.25.25.2000929
- Wagner GE, Föderl-Höbenreich E, Assig K, Lipp M, Berner A, Kohler C, Lichtenegger S, Stiehler J, Karoonboonyanan W, Thanapattarapairoj N, Promkong C, Koosakulnirand S, Chaichana P, Ehricht R, Gad AM, Söffing HH, Dunachie SJ, Chantratita N, Steinmetz I. (2020) Melioidosis DS rapid test: A standardized serological dipstick assay with increased sensitivity and reliability due to multiplex detection. PLoS Negl Trop Dis. 2020 Jul 13;14(7):e0008452. doi: 10.1371/journal.pntd.0008452
- El-Ashker M, Gwida M, Monecke S, Ehricht R, Elsayed M, El-Gohary F, Reißig A, Müller E, Paul A, Igbinosa EO, Beshiru A, Maurischat S. (2020) Microarray-based detection of resistance genes in coagulase-negative staphylococci isolated from cattle and buffalo with mastitis in Egypt. Trop Anim Health Prod. 2020 Nov;52(6):3855-3862. doi: 10.1007/s11250-020-02424-1
- Gawlik D, Ruppelt-Lorz A, Müller E, Reißig A, Hotzel H, Braun SD, Söderquist B, Ziegler-Cordts A, Stein C, Pletz MW, Ehricht R, Monecke S. (2020) Molecular investigations on a chimeric strain of Staphylococcus aureus sequence type 80. PLoS One. 2020 Oct 14;15(10):e0232071. doi: 10.1371/journal.pone.0232071
- Roberts MC, Joshi PR, Monecke S, Ehricht R, Müller E, Gawlik D, Diezel C, Braun SD, Paudel S, Acharya M, Khanal L, Koju NP, Chalise M, Kyes RC. (2020) Staphylococcus aureus and Methicillin Resistant S. aureus in Nepalese Primates: Resistance to Antimicrobials, Virulence, and Genetic Lineages. Antibiotics (Basel). 2020 Oct 13;9(10):689. doi: 10.3390/antibiotics9100689
- Senok A, Nassar R, Celiloglu H, Nabi A, Alfaresi M, Weber S, Rizvi I, Müller E, Reissig A, Gawlik D, Monecke S, Ehricht R. (2020) Genotyping of methicillin resistant Staphylococcus aureus from the United Arab Emirates. Nature Sci Rep. 2020 Oct 29;10(1):18551. doi: 10.1038/s41598-020-75565-w
- Mcmanus BA, Aloba BK, Earls MR, Brennan GI, O'Connell B, Monecke S, Ehricht R, Shore AC, Coleman DC.(2020) Multiple distinct outbreaks of Panton-Valentine leukocidin (PVL)-positive community-associated methicillin-resistant Staphylococcus aureus (CA-MRSA) in Ireland investigated by whole-genome sequencing. J Hosp Infect. 2020 Nov 28:S0195-6701(20)30545-4. doi: 10.1016/j.jhin.2020.11.021
- Monecke S, Syed MA, Khan MA, Ahmed S, Tabassum S, Gawlik D, Muller E, Reissig A, Braun SD, Ehricht R. Genotyping of methicillin-resistant Staphylococcus aureus from sepsis patients in Pakistan and detection of antibodies against staphylococcal virulence factors. Eur J Clin Microbiol Infect Dis. 2020;39(1):85-92. Epub 2019/09/05. doi: 10.1007/s10096-019-03695-9. PubMed PMID: 31482419.
- Sacco SC, Velazquez NS, Renna MS, Beccaria C, Baravalle C, Pereyra EAL, Monecke S, Calvinho LF, Dallard BE. Capacity of two Staphylococcus aureus strains with different adaptation genotypes to persist and induce damage in bovine mammary epithelial cells and to activate macrophages. Microb Pathog. 2020;142:104017. Epub 2020/02/02. doi: 10.1016/j.micpath.2020.104017. PubMed PMID: 32006636.
- Zurn K, Lander F, Hufnagel M, Monecke S, Berner R. Microarray Analysis of Group B Streptococci Causing Invasive Neonatal Early- and Late-onset Infection. Pediatr Infect Dis J. 2020;39(5):449-53. Epub 2020/02/25. doi: 10.1097/INF.0000000000002627. PubMed PMID: 32091495.
- Ahmed W, Neubauer H, Tomaso H, El Hofy FI, Monecke S, Abdeltawab AA, Hotzel H. Characterization of Staphylococci and Streptococci Isolated from Milk of Bovides with Mastitis in Egypt. Pathogens. 2020;9(5). Epub 2020/05/21. doi: 10.3390/pathogens9050381. PubMed PMID: 32429272; PubMed Central PMCID: PMCPMC7281669.
- Raafat D, Mrochen DM, Al'Sholui F, Heuser E, Ryll R, Pritchett-Corning KR, Jacob J, Walther B, Matuschka FR, Richter D, Westerhus U, Pikula J, van den Brandt J, Nicklas W, Monecke S, Strommenger B, van Alen S, Becker K, Ulrich RG, Holtfreter S. Molecular Epidemiology of Methicillin-Susceptible and Methicillin-Resistant Staphylococcus aureus in Wild, Captive and Laboratory Rats: Effect of Habitat on the Nasal S. aureus Population. Toxins (Basel). 2020;12(2). Epub 2020/01/30. doi: 10.3390/toxins12020080. PubMed PMID: 31991690; PubMed Central PMCID: PMCPMC7076793.
2019:
- Antók FI, Mayrhofer R, Marbach H, Masengesho JC, Keinprecht H, Nyirimbuga V, Fischer O, Lepuschitz S, Ruppitsch W, Ehling-Schulz M, Feßler AT, Schwarz S, Monecke S, Ehricht R, Grunert T, Spergser J, Loncaric I. (2020) Characterization of Antibiotic and Biocide Resistance Genes and Virulence Factors of Staphylococcus Species Associated with Bovine Mastitis in Rwanda. Antibiotics (Basel). 2019 Dec 18;9(1):1. doi: 10.3390/antibiotics9010001
- Bai X, Zhang J, Ambikan A, Jernberg C, Ehricht R, Scheutz F, Xiong Y, Matussek A: Molecular Characterization and Comparative Genomics of Clinical Hybrid Shiga Toxin-Producing and Enterotoxigenic Escherichia coli (STEC/ETEC) Strains in Sweden. Sci Rep 2019, 9(1):5619.
- Earls MR, Shore AC, Brennan GI, Simbeck A, Schneider-Brachert W, Vremera T, Dorneanu OS, Slickers P, Ehricht R, Monecke S et al: A novel multidrug-resistant PVL-negative CC1-MRSA-IV clone emerging in Ireland and Germany likely originated in South-Eastern Europe. Infection, genetics and evolution : journal of molecular epidemiology and evolutionary genetics in infectious diseases 2019, 69:117-126.
- Khan FA, Hellmark B, Ehricht R, Soderquist B, Jass J: Related carbapenemase-producing Klebsiella isolates detected in both a hospital and associated aquatic environment in Sweden. European journal of clinical microbiology & infectious diseases: official publication of the European Society of Clinical Microbiology 2018, 37(12):2241-2251.
- Krapf M, Muller E, Reissig A, Slickers P, Braun SD, Muller E, Ehricht R, Monecke S: Molecular characterisation of methicillin-resistant Staphylococcus pseudintermedius from dogs and the description of their SCCmec elements. Veterinary microbiology 2019, 233:196-203.
- Loncaric I, Kubber-Heiss A, Posautz A, Ruppitsch W, Lepuschitz S, Schauer B, Fessler AT, Krametter-Frotscher R, Harrison EM, Holmes MA et al: Characterization of mecC gene-carrying coagulase-negative Staphylococcus spp. isolated from various animals. Veterinary microbiology 2019, 230:138-144.
- Loreck K, Mitrenga S, Meemken D, Heinze R, Reissig A, Mueller E, Ehricht R, Engemann C, Greiner M: Development of a miniaturized protein microarray as a new serological IgG screening test for zoonotic agents and production diseases in pigs. PloS one 2019, 14(5):e0217290.
- Roberts MC, Fessler AT, Monecke S, Ehricht R, No D, Schwarz S: Molecular Analysis of Two Different MRSA Clones ST188 and ST3268 From Primates (Macaca spp.) in a United States Primate Center. Front Microbiol 2018, 9:2199.
- Weiss D, Gawlik D, Hotzel H, Engelmann I, Mueller E, Slickers P, Braun SD, Monecke S, Ehricht R: Fast, economic and simultaneous identification of clinically relevant Gram-negative species with multiplex real-time PCR. Future Microbiol 2019, 14:23-32.
- Pahlow S, Lehniger L, Hentschel S, Seise B, Braun SD, Ehricht R, Berg A, Popp J, Weber K: Rapid Isolation and Identification of Pneumonia-Associated Pathogens from Sputum Samples Combining an Innovative Sample Preparation Strategy and Array-Based Detection. ASC Omega 2019, 4(6):10362
- Coleman DC, Shore AC, Goering RV, Monecke S: Editorial: New Insights and Updates on the Molecular Epidemiology and Antimicrobial Resistance of MRSA in Humans in the Whole-Genome Sequencing Era. Front Microbiol 2019, 10:637.
- Dimitriou V, Biehl L, Hamprecht A, Vogel W, Dörfel D, Peter S, Schafhausen P, Rohde H, von Lilienfeld-Toal M, Klassert T et al: Controlling intestinal colonisation of high-risk haematology patients with ESBL-producing Enterobacteriaceae – a randomised, placebo controlled, multicentre, phase II trial (CLEAR). J Antimicrob Chemother 2019, dkz124, https://doi.org/10.1093/jac/dkz124.
- Hagel S, Makarewicz O, Hartung A, Weiss D, Stein C, Brandt C, Schumacher U, Ehricht R, Patchev V, Pletz MW: ESBL colonization and acquisition in a hospital population: The molecular epidemiology and transmission of resistance genes. PloS one 2019, 14(1):e0208505.
- Olofsson M, Matussek A, Ehricht R, Lindgren PE, Ostgren CJ: Differences in molecular epidemiology of Staphylococcus aureus and Escherichia coli in nursing home residents and people in unassisted living situations. J Hosp Infect 2019, 101(1):76-83.
- Thunberg U, Hugosson S, Fredlund H, Cao Y, Ehricht R, Monecke S, Mueller E, Engelmann S, Söderquist B: Anti-Staphylococcal humoral immune response in patients with chronic rhinosinusitis. Rhinology online 2019, 2:50-58.
- Tuchscherr L, Pollath C, Siegmund A, Deinhardt-Emmer S, Hoerr V, Svensson CM, Thilo Figge M, Monecke S, Loffler B: Clinical S. aureus Isolates Vary in Their Virulence to Promote Adaptation to the Host. Toxins (Basel) 2019, 11(3).
- Weiss D, Gawlik D, Hotzel H, Engelmann I, Mueller E, Slickers P, Braun SD, Monecke S, Ehricht R: Fast, economic and simultaneous identification of clinically relevant Gram-negative species with multiplex real-time PCR. Future Microbiol 2019, 14:23-32.